|
تضامنًا مع حق الشعب الفلسطيني |
التكرار المقلوب
هذه مقالة غير مراجعة. (ديسمبر 2021) |
التكرار المقلوب (بالإنجليزية:Inverted repeat (IR))هوعبارة عن تسلسل واحد من قطع النوكليوتيدات المتبوعة بالاسفل من خلال مكملاتها العكسية[1] . يمكن أن يكون التسلسل المتداخل للنيوكليوتيدات بين التسلسل الأولي والمكمل العكسي بأي طول بما في ذلك الصفر. على سبيل المثال 5'---TTACGnnnnnnCGTAA---3' عبارة عن تسلسل تكرار مقلوب. عندما يكون الطول التداخل صفراً، يكون التسلسل المركب عبارة عن تسلسل متناظر[2].
تشكل كل من التكرارات المقلوبة والتكرارات المباشرة أنواعًا من تسلسل النوكليوتيداتالتي تحدث بشكل متكرر. تسلسلات الحمض النووي المتكررة غالبًا ما تتروح هذهِ من زوج نيوكليوتيدات إلى جين كامل، في حين أن تقارب التسلسلات المتكررة يختلف بين الصفائف المترادفة المتناثرة والبسيطة[3]. تسلسل التضاعف الترادفي القصير قد يكون موجود فقط كنسخ قليلة في منطقة صغيرة إلى آلاف النسخ المتناثرة في جميع أنحاء الجينوم في معظم الخلايا حقيقية النوى.[4] المتسلسلات المتكررة مع تقريبا 10-100 زوج قاعدي تُعرف باسم المتسلسلات الصغيرة (بالإنجليزية: minisatellites)، في حين تُعرف المتسلسلات المتكررة القصيرة التي تحتوي في الغالب على 2-4 أزواج قاعدية بالسواتل الصغرى (بالإنجليزية: microsatellites).[5] التكرار الأكثر شيوعا تشمل تكرار ثنائي النوكليوتيد، التي لديها قواعد AC على شريط الحمض النووي واحد، و GT على الشريط التكميلي. بعض عناصر الجينوم ذات تسلسل فريد تعمل كإكسونات، وإنترونات، وDNA منظم.على الرغم من أن أكثر المواقع الشائعة للتسلسل المتكرر هي سنترومير(قسيم مركزي)، والتلومير (قسيم طرفي)[6] ، جزء كبير من التسلسلات المتكررة موجودة في الجينوم وسط DNA الغيرمشفر.[5]
التكرار المقلوب له عدد من الوظائف البيولوجية الهامة. فهي تحدد الحدود في الترانسبوزونات(الترانسبوزونات: (جين قافزة)هو تسلسل الحمض النووي الذي يتحرك حول مواقع مختلفة داخل الجينوم) وتشير إلى المناطق القادرة على التكامل الذاتي بين ازواج القواعد (المناطق ضمن تسلسل واحد التي يمكن أن تقوم على أساس زوج مع بعضها البعض.). وتلعب هذه الخواص دورا هاما في عدم استقرار[7] الجينوم وتساهم ليس فقط في التطورالخلايا والتنوع الجيني[8] ولكن أيضا في الطفرة والمرض[9]. لدراسة هذه الآثار بالتفصيل، تم وضع عدد من البرامج وقواعد البيانات للمساعدة في اكتشاف وشرح التكرار المقلوب في مختلف الجينوم.
فهم التكرارات المقلوبة

مثال على التكرار المقلوب
نبدأ بهذا التسلسل الأولي:
5'-TTACG-3‘
المكمل الذي تم إنشاؤه بواسطة الازدواج القاعدي هو:
3'-AATGC-5‘
التكملة العكسيّة هي:
5'-CGTAA-3‘
وتسلسل التكرار المقلوب هو:
5'---TTACGnnnnnnCGTAA---3‘
“nnnnnn" يمثل أي عدد من النوكليوتيدات المتدخلة.
التكرار المباشر
التكرار المباشر يحدث عندما يتكرر تسلسل مع نفس النمط اخر المسار السفلي.
5´ TTACGnnnnnnTTACG 3´
3´ AATGCnnnnnnAATGC 5´
التضاعف الترادفي
التكرار المباشر مع عدم وجود نيوكليوتيدات متداخلة بين التسلسل الأولي واخر المسار السفلي هو تكرار ترادفي. يشير تسلسل النوكليوتيدات المكتوب بأحرف غامقة إلى التسلسل المتكرر.
5´ TTACGTTACG 3´
3´ AATGCAATGC 5´
التسلسلات المتناظرة
إن تسلسل التكرار المقلوب مع عدم وجود نوكليوتيدات متدخلة بين التسلسل الأولي والتكملة العكسية للمسار السفلي هو عبارة عن تسلسل متناظر[1].
مثال:
الخطوة 1: ابدأ بتكرارالمقلوب:
5' TTACGnnnnnnCGTAA 3‘
الخطوة 2: حذف النوكليوتيدات المتدخلة:
5' TTACGCGTAA 3‘
هذا التسلسل الناتج متناوب لأنه مكمل عكسي لنفسه.[1]
5' TTACGCGTAA 3'
تسلسل الاختبار (من الخطوة 2 مع إزالة النيوكليوتيدات المتداخلة)
3' AATGCGCATT 5'
تكملة تسلسل الاختبار
5' TTACGCGTAA 3‘
تكملة عكسية، هذا هو نفس تسلسل الاختبار أعلاه، وبالتالي فهو متناظر.
السمات والوظائف البيولوجية
الظروف التي تفضل التكوين
تُستمد التكرارات المتنوعة على نطاق الجينوم من عناصر نَقُولة، والتي تُفهم الآن على أنها «قفزة» حول مواقع جينومية مختلفة، دون نقل نسخها الأصلية.[10] ويضمن إغلاق نفس التسلسل في وقت لاحق على مدى أجيال عديدة تعددها في جميع أنحاء الجينوم.[10] إن عملية إعادة التركيب المحدودة لتسلسل بين عنصرين متميزين للتتابع والمعروفين باسم إعادة التركيب الخاصة بالموقع (CSSR) تؤدي إلى انعكاسات لجزء DNA ، استناداً إلى ترتيب تسلسل التعرف على إعادة التتابع على DNA المتبرع وDNA المستقبل. [10] ومرة أخرى، فإن اتجاه اثنين من مواقع إعادة التقسيم داخل DNA المتبرع نسبة إلى عدم تماثل تسلسلاتDNA المتدخلة، المعروفة باسم منطقة التقاطع، هو أمر محوري لتكوين أي من التكرارات المقلوبة أو التكرارات المباشرة.[10] وبالتالي، فإن إعادة التشكيل التي تحدث في زوج من المواقع المقلوبة سوف تعكس تسلسلDNA بين الموقعين.[10] وقد لوحظت كروموسومات مستقرة جدا مقارنة مع أعداد أقل نسبيا من التكرار المقلوب من التكرار المباشر، مما يشير إلى وجود علاقة بين استقرار الكروموسوم وعدد التكرار.[11]
المناطق التي يكون الوجود فيها إلزاميًا
وقد لوحظت التكرارات المقلوبة الطرفية في DNA لمختلف الترانسبوسونات حقيقية النواة، على الرغم من أن مصدرها لا يزال غير معروف[12]. التكرار المقلوب موجود بشكل رئيسي في أصول تكرار عضو الخلية والأعضاء التي تتراوح من عاثية البلازميد، الميتوكوندريا، وفيروسات حقيقية النواة إلى خلايا الثدييات[13]. اصول النسخ المتماثل (التكرار) للعاثية G4 والعاثيات الأخرى ذات الصلة تشمل قطع من ما يقارب 139 قاعدة نوكليوتيد التي تشمل ثلاثة تكراراتمقلوبة التي هي ضرورية للتكرار التمهيدي. [13]
في الجينوم
إلى حد كبير، وكثيرا ما يلاحظ وجود أجزاء من تكرارات النوكليوتيد كجزء من تركيبة نادرة من DNA[14] . التكرارات الثلاثة الرئيسية التي توجد إلى حد كبير في بنيات DNA معينة تشمل التكرارات المقلوبة الهوموبورين-هوموبيريميدين الدقيقة، حدوث شائع في المطابقات H الحلزونية الثلاثية التي قد تشتمل على ثلاثيات نيوكليوتيدات TAT أو CGC. يمكن وصف البقية على أنها تكرارات مقلوبة طويلة بها انحناء لإنتاج كشكل دبابيس الشعر وصليب الشكل، وأخيراً التكرارات الترادفية المباشرة، والتي توجد عادةً في الهياكل الموصوفة بأنها Z-DNA ذات الحلقة المنزلقة والصليبية الشكل واليسرى.[14]
شائع في مختلف الكائنات

تشير الدراسات السابقة إلى أن التكرار هو سمة مشتركة من حقيقيات النوى على عكس بدائيات النوى وعتائق.[14] تشير تقارير أخرى إلى أنه بغض النظر عن النقص المقارن لعناصر التكرار في جينومات بدائية النواة، فإنها مع ذلك تحتوي على مئات أو حتى آلاف التكرارات الكبيرة.[15] يبدو أن التحليل الجيني الحالي يشير إلى وجود فائض كبير من التكرارات المقلوبة المتقنة في العديد من الجينومات بدائية النواة مقارنة بجينومات حقيقية النواة.[16]
لتحديد ومقارنة التكرارات المقلوبة بين عدة أنواع، وبالتحديد على العتائق، انظر [17]
التكرار المقلوب في العقد الزائفة
العقد الزائفة (العقد الكاذبة) هي تتابعات تنظيمية هيكلية شائعة موجودة في RNA . وهي تتكون من حلقتين جذعيتين مزدوجتين بحيث يتكون جذع هيكل واحد من حلقة الآخر. هناك العديد من الطوبولوجيا القابلة للطي بين العقد الزائفة وتباين كبير في أطوال الحلقة، مما يجعلها مجموعة متنوعة هيكليًا.[18]
التكرارات المقلوبة هي مكون رئيسي للعقدة الزائفة كما يمكن رؤيته في الرسم التوضيحي للعقدة الزائفة التي تحدث بشكل طبيعي الموجودة في مكون التيلوميراز RNA البشري[19].أربعة مجموعات مختلفة من التكرارات المقلوبة تشارك في هذا الهيكل. المجموعات 1 و 2 هي جذع الحلقة الجذعية A وهي جزء من الحلقة الجذعية B .وبالمثل، فإن المجموعتين 3 و4 هما جذع الحلقة الجذعية B وجزء من الحلقة الجذعية A .
تلعب العقدة الكاذبة عددًا من الأدوار المختلفة في علم الأحياء. إن عقدة التيلوميراز الكاذبة في الرسم التوضيحي ضرورية لنشاط هذا الإنزيم.[19] ينثني الريبوزيم لفيروس دلتا الكبد (HDV) في بنية العقدة الكاذبة المزدوجة ويشق ذاتيًا جينومه الدائري RNA أحادي الجينوم. تلعب العقد الكاذبة أيضًا دورًا في طَفْرَةُ انْزِياحِ الرايبوزوم المبرمجة الموجود في بعض الفيروسات والمطلوبة في تكرار الفيروسات القهقرية (الرجعية).[18]
في الريبوسويتشات
التكرار المقلوب يلعب دورا هاما في الريبوسويتشات، وهي العناصر التنظيمية للRNA التي تتحكم في التعبير عن الجينات التي تنتج mRNA ، والتي هي جزء منها. [10]يظهر مثال مبسط لريبوسويتش أحادي النوكليوتيد الفلافين (FMN) في الرسم التوضيحي. يوجد هذا الريبوسويتش في نسخة mRNA وله العديد من هياكل الحلقة الجذعية أعلى المنطقة المشفرة. ومع ذلك، يتم عرض الحلقات الجذعية فقط في الرسم التوضيحي، والذي تم تبسيطه بشكل كبير للمساعدة في إظهار دور التكرارات المقلوبة. يوجد تكرار مقلوب متعدد في هذا الريبوسويتش كما هو موضح باللون الأخضر (خلفية صفراء) والأزرق (خلفية برتقالية).

في حالة عدم وجود FMN ، فإن البنية المضادة للإنهاء هي الشكل المفضل لنسخة mRNA. يتم إنشاؤه عن طريق ازدواج القواعد لمنطقة التكرارالمقلوب المحاطة بدائرة باللون الأحمر. عندما تكون FMN موجودة، قد ترتبط بالحلقة وتمنع تشكيل الهيكل المانع للإنهاء. يسمح هذا لمجموعتين مختلفتين من التكرارات المقلوبة لأزدواج القواعد وتشكيل بنية الإنهاء[20]. الحلقة الجذعية على الطرف ' 3 هي نهاية نسخ لأن التسلسل الذي يليها مباشرة عبارة عن سلسلة من اليورسيل (U).إذا تشكلت هذه الحلقة الجذعية (بسبب وجود FMN) مع ظهور شريط RNA المتزايد من RNA بوليمراز المعقد، فسيخلق توترًا هيكليًا كافيًا لإحداث فصل RNA وبالتالي إنهاء النسخ. يحدث التفكك بسهولة لأن الاقتران الأساسي بينU's في RNAو A's يحدث التفكك بسهولة لأن ازدواج القواعد بين U's في RNA و A في شريط القالب هي الأضعف بين جميع أزواج القواعد[10]. وبالتالي، عند مستويات تركيز أعلى، FMN تنظم أسفل نسخها الخاص عن طريق زيادة تشكيل بنية الإنهاء.
الطفرات والمرض
غالبًا ما توصف التكرارات المقلوبة بأنها «المناطق الشديدة التأثر» لعدم الاستقرار الجيني لحقيقيات النواة وبدائية النواة. [7]يُعتقد أن التكرارات المقلوبة الطويلة تؤثر بشكل كبير على استقرار جينوم الكائنات الحية المختلفة.[21] يتجلى هذا في بكتيريا الE. coli ، حيث نادرًا ما يتم تكرار التسلسلات الجينومية ذات التكرارات الطويلة المقلوبة، بل يتم حذفها بسرعة.[21] مرة أخرى، التكرارات المقلوبة الطويلة التي لوحظت في الخميرة تفضل بشكل كبير إعادة التركيب داخل نفس الكروموسومات المجاورة، مما يؤدي إلى معدل حذف مرتفع للغاية.[21] أخيرًا، لوحظ أيضًا معدل عالٍ جدًا من الحذف وإعادة التركيب في مناطق كروموسومات الثدييات ذات التكرارات المقلوبة.[21] أخيرًا، لوحظ أيضًا معدل عالٍ جدًا من الحذف وإعادة التركيب في مناطق كروموسومات الثدييات ذات التكرارات المقلوبة.[21] الاختلافات المبلغ عنها في استقرار جينومات الكائنات الحية المترابطة هي دائمًا مؤشر على وجود تباين في التكرارات المقلوبة.[11] وينتج عدم الاستقرار عن ميل التكرار المقلوب إلى الطي في هياكل DNA المشابهة للشعر أو الصلبة.[7] وبالتالي، تؤدي التكرارات المقلوبة إلى تكوينات خاصة في كل من RNA وDNA التي يمكن أن تسبب في النهاية الطفرات والمرض.[9]

يُظهر الرسم التوضيحي تكرارًا مقلوبًا يخضع لعملية قذف صليبي الشكل. يتكررDNA في منطقة التكرار المقلوب ثم يعيد الاتحاد، مكونًا تقاطعًا رباعي الاتجاهات بهيكلان من حلقة جذعية. تحدث البنية الصليبية لأن التسلسلات المتكررة المقلوبة تتزاوج ذاتيًا مع بعضها البعض على شريطها الخاص.[22]
يمكن أن تؤدي الأشكال الصليبية المبثوقة إلى حدوث طفرة انزِياح الإِطارعندما يكون تسلسل DNA قد انعكس على التكرارات في شكل تسلسل متناظر مدمج مع مناطق التكرارالمباشر على كلا الجانبين. أثناء النسخ، يمكن أن يؤدي الانزلاق والتفكك الجزئي للبوليميراز من شريط القالب إلى حدوث طفرات في الحذف والإدراج.[9] يحدث الحذف عندما يشكل جزء من شريط القالب غير الملفوف حلقة جذعية يتم «تخطيها» بواسطة آلية النسخ. يحدث الإدراج عندما تتشكل حلقة جذعية في جزء منفصل من الشريط الناشئ (المركب حديثًا) مما يتسبب في نسخ جزء من شريط القالب مرتين.[9]
نقص مضاد الثرومبين من طفرة نقطية
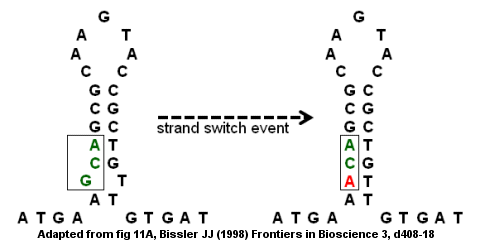
يمكن أن تؤدي التكرارات المقلوبة غير السليمة إلى حدوث طفرات من خلال التبديل داخل الشريط وداخل الشريط.[9] منطقة ترميز جينات مضاد الثرومبين الثالث هي مثال على التكرار المقلوب غير السليم (غير المكتمل) كما هو موضح في الشكل على اليمين. يتشكل بنية الحلقة الجذعية مع نتوء في الأسفل لأن الحرفين G و T لا يتزاوجان. يمكن أن يؤدي حالة تبديل شريط في G (في النتوء) إلى الاستبدال بـ A الذي يزيل «الاضطراب» في التكرار المقلوب ويوفر بنية حلقة جذعية أقوى. ومع ذلك، فإن الاستبدال يخلق أيضًا طفرة نقطية تقوم بتحويل كودون GCA إلى ACA. إذا تبع حالة تبديل الشريط بجولة ثانية من تضاعف DNA ، فإن الطفرة قد تصبح دائمة في الجينوم وتؤدي إلى المرض. على وجه التحديد، الطفرة المُغلطة قد تؤدي إلى خلل في الجين ونقص في مضاد الثرومبين مما قد يؤدي إلى تطور الجلطات الدموية الوريدية (جلطات الدم داخل الوريد).[9]

تكوين العظام الناقص من طفرة انزياح الإطار
يمكن أن تؤدي الطفرات في جين الكولاجين إلى الإصابة بمرض تكوين العظام الناقص، والذي يتميز بهشاشة العظام[9]. تؤدي إضافة T إلى إنشاء «تطابق» زوج القواعد مع الأدينين (A) الذي كان سابقًا «نتوءًا» على الجانب الأيسر من الجذع. في حين أن هذه الإضافة تجعل الجذع أقوى وتحسن التكرار المقلوب، فإنها تخلق أيضًا طفرة الإزاحة في تسلسل النيوكليوتيدات التي تغير إطار القراءة وستؤدي إلى تعبير غير صحيح عن الجين.[9]
البرامج وقواعد البيانات
توفر القائمة التالية معلومات وروابط خارجية لمختلف البرامج وقواعد البيانات للتكرارات المقلوبة:
- non-B DB قاعدة بيانات للتعليقات التوضيحية المتكاملة وتحليل الزخارف المكونة لِ non-B DNA.[23] يتم توفير قاعدة البيانات هذه من قبل مركز الحوسبة الطبية الحيوية المتقدمة (ABCC) في مختبر فريدريك الوطني لأبحاث السرطان (FNLCR). وهي تغطي مطابقة A-DNA وZ-DNA المعروفة باسم "DNA non-B" لأنها ليست أكثر أشكال B-DNA شيوعًا للحلزون المزدوج Watson-Crick الأيمن.وتشمل هذه "non-B DNAs" هياكل Z-DNA اليسرى، والصليبية الشكل، والثلاثية، ورباعية الأطراف، وهياكل دبوس الشعر.[23] يمكن إجراء عمليات البحث على مجموعة متنوعة من «الأنواع المكررة» (بما في ذلك التكرارات المقلوبة) وعلى عدة أنواع.
- Inverted Repeats Database جامعة بوسطن . قاعدة البيانات هذه هي تطبيق ويب يسمح بالاستعلام عن عمليات التكرار وتحليلها في مشروع قاعدة البيانات العامة. يمكن للعلماء أيضًا تحليل التسلسلات الخاصة بهم باستخدام خوارزمية الباحث عن التكرار المقلوب.[24]
- P-MITE: a Plant MITE database تحتوي قاعدة البيانات هذه الخاصة بالعناصر القابلة للتحويل المقلوب المتكرر المصغر (MITEs) على تسلسلات من جينومات النباتات. قد يتم البحث عن التسلسلات أو تنزيلها من قاعدة البيانات.[25]
- EMBOSS هي «مجموعة برامج البيولوجيا الجزيئية الأوروبية المفتوحة» التي تعمل على أنظمة تشغيل شبيهة بـ UNIX وUNIX.[26] تتوفر ملفات التوثيق والبرامج المصدر على EMBOSS website . التطبيقات المتعلقة بشكل خاص بالتكرارات المقلوبة مذكورة أدناه:
- EMBOSS einverted : تجد التكرارات المقلوبة في تسلسل النيوكليوتيدات. يمكن تعيين قيم البدء للحد من نطاق البحث.[26]
- EMBOSS palindrome: تجد السلاسل المتناظرة مثل مناطق الحلقة الجذعية في تسلسل النيوكليوتيدات. سيجد البرنامج تسلسلات تتضمن أقسامًا من عدم التطابق والفجوات التي قد تتوافق مع الانتفاخات في حلقة جذعية.[26]
المراجع
- ^ أ ب ت David W.؛ Wassenaar، Trudy M.؛ Borini، Stefano. Word Frequencies and Repeats. London: Springer London. ص. 137–150. ISBN:978-1-84800-254-8. مؤرشف من الأصل في 2021-12-27.
- ^ Ye, Congting; Ji, Guoli; Li, Lei; Liang, Chun (19 Nov 2014). Tosatto, Silvio C. E. (ed.). "detectIR: A Novel Program for Detecting Perfect and Imperfect Inverted Repeats Using Complex Numbers and Vector Calculation". PLoS ONE (بEnglish). 9 (11): e113349. DOI:10.1371/journal.pone.0113349. ISSN:1932-6203. Archived from the original on 2021-05-18.
- ^ Richards, GR; Richards, RI (Apr 25, 1995). "Simple tandem DNA repeats and human genetic disease". Proceedings of the National Academy of Sciences of the United States of America. 92 (9): 3636–41. Bibcode:1995PNAS...92.3636S. doi:10.1073/pnas.92.9.3636. PMC 42017. PMID 7731957.
- ^ van Belkum, A; Scherer, S; van Alphen, L; Verbrugh, H (June 1998). "Short-sequence DNA repeats in prokaryotic genomes". Microbiology and Molecular Biology Reviews. 62 (2): 275–93. doi:10.1128/MMBR.62.2.275-293.1998. PMC 98915. PMID 9618442.
- ^ أ ب Ramel, C (June 1997). "Mini- and microsatellites". Environmental Health Perspectives. 105 Suppl 4 (Suppl 4): 781–9. doi:10.2307/3433284. JSTOR 3433284. PMC 1470042. PMID 9255562.
- ^ Eichler, EE (August 1998). "Masquerading repeats: paralogous pitfalls of the human genome". Genome Research. 8 (8): 758–62. doi:10.1101/gr.8.8.758. PMID 9724321.
- ^ أ ب ت Mirkin, I; Narayanan, V; Lobachev, KS; Mirkin, SM (Jul 22, 2008). "Replication stalling at unstable inverted repeats: interplay between DNA hairpins and fork stabilizing proteins". Proceedings of the National Academy of Sciences of the United States of America. 105 (29): 9936–41. Bibcode:2008PNAS..105.9936V. doi:10.1073/pnas.0804510105. PMC 2481305. PMID 18632578.
- ^ Lin, CT; Lin, WH; Lyu, YL; Whang-Peng, J (Sep 1, 2001). "Inverted repeats as genetic elements for promoting DNA inverted duplication: implications in gene amplification". Nucleic Acids Research. 29 (17): 3529–38. doi:10.1093/nar/29.17.3529. PMC 55881. PMID 11522822.
- ^ أ ب ت ث ج ح خ د Bissler, JJ (Mar 27, 1998). "DNA inverted repeats and human disease" (PDF). Frontiers in Bioscience. 3 (4): d408–18. doi:10.2741/a284. PMID 9516381. S2CID 12982. Archived from the original (PDF) on March 3, 2019.
- ^ أ ب ت ث ج ح خ Molecular biology of the gene (ط. Seventh edition). Boston. 2014. ISBN:978-0-321-76243-6. OCLC:824087979. مؤرشف من الأصل في 2021-09-29.
{{استشهاد بكتاب}}:|طبعة=يحتوي على نص زائد (مساعدة)صيانة الاستشهاد: مكان بدون ناشر (link) - ^ أ ب Achaz, G; Coissac, E; Netter, P; Rocha, EP (August 2003). "Associations between inverted repeats and the structural evolution of bacterial genomes". Genetics. 164 (4): 1279–89. doi:10.1093/genetics/164.4.1279. PMC 1462642. PMID 12930739.
- ^ Zhang, HH; Xu, HE; Shen, YH; Han, MJ; Zhang, Z (January 2013). "The Origin and Evolution of Six Miniature Inverted-Repeat Transposable Elements in Bombyx mori and Rhodnius prolixus". Genome Biology and Evolution. 5 (11): 2020–31. doi:10.1093/gbe/evt153. PMC 3845634. PMID 24115603.
- ^ أ ب Pearson, CE; Zorbas, H; Price, GB; Zannis-Hadjopoulos, M (October 1996). "Inverted repeats, stem-loops, and cruciforms: significance for initiation of DNA replication". Journal of Cellular Biochemistry. 63 (1): 1–22. doi:10.1002/(SICI)1097-4644(199610)63:1<1::AID-JCB1>3.0.CO;2-3. PMID 8891900. S2CID 22204780.
- ^ أ ب ت Heringa, Jaap (1998-06). "Detection of internal repeats: how common are they?". Current Opinion in Structural Biology (بEnglish). 8 (3): 338–345. DOI:10.1016/S0959-440X(98)80068-7. Archived from the original on 30 يونيو 2018.
{{استشهاد بدورية محكمة}}: تحقق من التاريخ في:|تاريخ=(help) - ^ Treangen, TJ; Abraham, AL; Touchon, M; Rocha, EP (May 2009). "Genesis, effects and fates of repeats in prokaryotic genomes" (PDF). FEMS Microbiology Reviews. 33 (3): 539–71. doi:10.1111/j.1574-6976.2009.00169.x. PMID 19396957.
- ^ Ladoukakis, ED; Eyre-Walker, A (September 2008). "The excess of small inverted repeats in prokaryotes" (PDF). Journal of Molecular Evolution. 67 (3): 291–300. Bibcode:2008JMolE..67..291L. CiteSeerX 10.1.1.578.7466. doi:10.1007/s00239-008-9151-z. PMID 18696026. S2CID 29953202.
- ^ Hosseini, M; Pratas, D; Pinho, AJ (2017). "On the Role of Inverted Repeats in DNA Sequence Similarity". 11th International Conference on Practical Applications of Computational Biology & Bioinformatics. Advances in Intelligent Systems and Computing. 616. Springer. pp. 228–236. doi:10.1007/978-3-319-60816-7_28. ISBN 978-3-319-60815-0.
- ^ أ ب Staple, DW; Butcher, SE (June 2005). "Pseudoknots: RNA structures with diverse functions". PLOS Biology. 3 (6): e213. doi:10.1371/journal.pbio.0030213. PMC 1149493. PMID 15941360
- ^ أ ب Chen, JL; Greider, CW (Jun 7, 2005). "Functional analysis of the pseudoknot structure in human telomerase RNA". Proceedings of the National Academy of Sciences of the United States of America. 102 (23): 8080–5, discussion 8077–9. Bibcode:2005PNAS..102.8080C. doi:10.1073/pnas.0502259102. PMC 1149427. PMID 15849264.
- ^ Winkler, WC; Cohen-Chalamish, S; Breaker, RR (Dec 10, 2002). "An mRNA structure that controls gene expression by binding FMN". Proceedings of the National Academy of Sciences of the United States of America. 99 (25): 15908–13. Bibcode:2002PNAS...9915908W. doi:10.1073/pnas.212628899. PMC 138538. PMID 12456892.
- ^ أ ب ت ث ج Stormo, G; Chang, KY; Varley, K; Stormo, GD (Feb 28, 2007). Hall, Neil (ed.). "Evidence for active maintenance of inverted repeat structures identified by a comparative genomic approach". PLOS ONE. 2 (2): e262. Bibcode:2007PLoSO...2..262Z. doi:10.1371/journal.pone.0000262. PMC 1803023. PMID 17327921
- ^ Ramreddy, T; Sachidanandam, R; Strick, TR (May 2011). "Real-time detection of cruciform extrusion by single-molecule DNA nanomanipulation". Nucleic Acids Research. 39 (10): 4275–83. doi:10.1093/nar/gkr008. PMC 3105387. PMID 21266478.
- ^ أ ب Cer, RZ; Donohue, DE; Mudunuri, US; Temiz, NA; Loss, MA; Starner, NJ; Halusa, GN; Volfovsky, N; Yi, M; Luke, BT; Bacolla, A; Collins, JR; Stephens, RM (January 2013). "Non-B DB v2.0: a database of predicted non-B DNA-forming motifs and its associated tools". Nucleic Acids Research. 41 (Database issue): D94–D100. doi:10.1093/nar/gks955. PMC 3531222. PMID 23125372.
- ^ Gelfand, Y; Rodriguez, A; Benson, G (January 2007). "TRDB--the Tandem Repeats Database". Nucleic Acids Research. 35 (Database issue): D80–7. doi:10.1093/nar/gkl1013. PMC 1781109. PMID 17175540 "نسخة مؤرشفة". مؤرشف من الأصل في 2021-09-22. اطلع عليه بتاريخ 2021-12-24.
{{استشهاد ويب}}: صيانة الاستشهاد: BOT: original URL status unknown (link) - ^ Chen, J; Hu, Q; Zhang, Y; Lu, C; Kuang, H (Oct 29, 2013). "P-MITE: a database for plant miniature inverted-repeat transposable elements". Nucleic Acids Research. 42 (1): D1176–81. doi:10.1093/nar/gkt1000. PMC 3964958. PMID 24174541.
- ^ أ ب ت Rice, P; Longden, I; Bleasby, A (June 2000). "EMBOSS: the European Molecular Biology Open Software Suite". Trends in Genetics. 16 (6): 276–7. doi:10.1016/S0168-9525(00)02024-2. PMID 10827456.